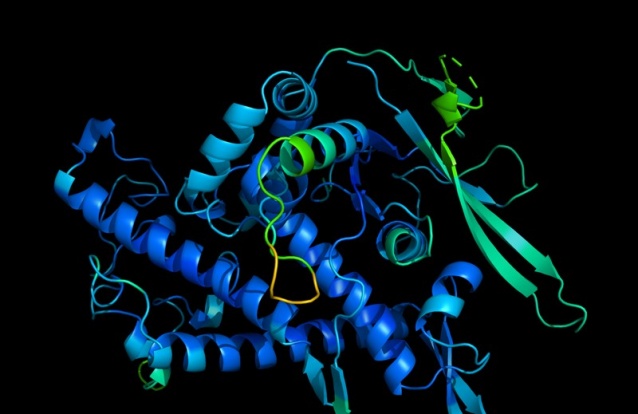
Além do Gemini, a Google também tem vindo a trabalhar no desenvolvimento de soluções de Inteligência Artificial para a área da Ciência. O AlphaFold é uma destas soluções e já revolucionou o estudo das proteínas. Agora, Demis Hassabis, responsável de IA da Google e CEO da DeepMind, revela que o software foi melhorado significativamente e que consegue modelar outras moléculas, incluindo ADN, e as interações entre os anticorpos produzidos pelo sistema imunitário e as moléculas de organismos doentes.
O avanço no AlphaFold 3 foi possível graças a algumas técnicas usadas e testadas em geradores de imagens baseados em IA, como um modelo de difusão, que refina as estruturas moleculares que o software produz. “Isto é exatamente o que precisamos para descobrir novos medicamentos: ver como uma pequena molécula vai interagir com um medicamento, quão forte será a ligação e a que outros compostos se pode ligar”, realça Demis Hassabis, citado pela Wired.
O algoritmo será capaz de modelar grandes moléculas, como o ADN e o RNA, mas também outras mais pequenas, como os iões de metal, prevendo com grande precisão como interagem umas com as outras, noticia a revista, citando o estudo publicado pela Google.
O trabalho no AlphaFold 3 foi feito entre a Google DeepMind e a Isomorphic Labs, uma empresa que também é liderada por Demis Hassabis. Esta versão vai ser disponibilizada gratuitamente aos investigadores via cloud, mas o software não vai ser de código aberto, ao contrário das versões anteriores do AlphaFold.
Até há alguns anos, o estudo das estruturas de proteínas implicava o uso de microscópios e uma técnica de raios-X conhecida por cristalografia. No entanto, investigadores começaram a aplicar mais tarde técnicas de deep learning, que estão na base de muitos dos avanços da IA neste tipo de estudos.
Em 2018, a DeepMind revelou estar a desenvolver o AlphaFold para prever a forma das proteínas com maior precisão. Em 2020, o AlphaFold 2 apresentou resultados convincentes que entusiasmaram os investigadores. Um ano depois surgiu a versão de código aberto do software que pode ser usada gratuitamente, incluindo 350 mil estruturas de proteínas previstas. Em 2022, o número de estruturas aumentou para mais de dois milhões.